Explorons le code source du vaccin BioNTech / Pfizer contre le SARS-CoV-2
December 27,
2020
Traduction
par Renaud
Guérin de l’article original de Bert Hubert
Merci
à Mathieu
Rebeaud et Genetix pour la relecture et les
corrections !
Mise
à jour 1/1/2021 : la suite de l’article est en ligne.
Bienvenue
!
Dans
cet article, nous allons déchiffrer caractère après caractère le code source du
vaccin SARS-CoV-2 à ARN messager (ARNm) de BioNTech / Pfizer.
Cette
phrase peut vous interloquer : après tout, un vaccin est un liquide injecté
dans votre bras. Alors, comment peut-on parler de code source?
C’est
une bonne question : commençons à y répondre en jetant un œil à une petite
partie de ce code source du vaccin BioNTech / Pfizer, nom de code BNT162b2,
alias Tozinameran ou encore Comirnaty.

Les
500 premiers nucléotides (caractères) de l’ARNm BNT162b2. Source: Organisation mondiale de la santé.
Ce
code numérique est au cœur du vaccin ARNm BNT162b. Il fait 4284 caractères de
long (ce qui tiendrait en quelques dizaines de tweets).
Au
commencement du processus de production du vaccin, quelqu’un a téléchargé ce
code dans une imprimante à ADN (oui, ça existe), qui a produit des molécules
d’ADN bien réelles à partir de ces données numériques.

Une
imprimante à ADN Codex
DNA BioXp 3200
Cette
machine sort de minuscules quantités d’ADN qui, après de nombreux traitements
biologiques et chimiques, finissent sous forme d’ARN (on en reparle plus tard)
dans un flacon de vaccin. Une dose de 30 microgrammes contient de fait 30
microgrammes d’ARN. Il y a en plus un système d’enrobage lipidique (gras)
plutôt malin qui permet à l’ARNm de pénétrer dans nos cellules.
L’ARN
est la version «mémoire de travail volatile» de l’ADN. L’ADN, c’est la clé USB
de la biologie : il est très durable, très fiable et a de la redondance
interne. Mais de la même manière que les ordinateurs n’exécutent pas de code
directement à partir d’une clé USB, là aussi le code est copié en premier lieu
dans un système plus rapide, plus polyvalent, mais aussi beaucoup plus fragile.
En
informatique, c’est la mémoire vive (RAM). En biologie, c’est l’ARN. La
ressemblance est frappante : contrairement à la mémoire flash, le contenu de la
mémoire vive se dégrade très rapidement, à moins d’être constamment rafraîchi.
La raison pour laquelle le vaccin à ARNm Pfizer / BioNTech doit être stocké
dans des congélateurs de compétition est la même: l’ARN est une fleur fragile.
Chaque
caractère d’ARN pèse de l’ordre de 0,53 · 10⁻²¹ gramme, ce qui signifie qu’il y a 6 · 10¹⁶ caractères dans une seule dose de vaccin de 30 microgrammes.
Exprimé
en octets, cela représente environ 25 Po (pétaoctets), même s’il faut préciser
qu’il s’agit d’environ 2000 milliards de répétitions des mêmes 4284 caractères.
Le contenu informationnel réel du vaccin est d’un peu plus d’un kilo-octet. Legénome du SARS-CoV-2 en lui-même pèse
environ 7,5 kilo-octets.
Un tout petit peu de contexte
L’ADN
est un code numérique. Contrairement aux ordinateurs qui utilisent 0 et 1, la
vie utilise A, C, G et U / T (les «nucléotides», «nucléosides» ou «bases»).
Dans
les ordinateurs, le 0 ou le 1 sont stockés sous forme d’une charge (ou d’une
absence de charge), d’un courant, d’une transition magnétique, d’une tension,
d’une modulation de signal, d’un changement de réflexivité… En d’autres termes,
ces 0 et 1 ne sont pas qu’un concept abstrait : ils prennent la forme d’électrons
et de nombreuses autres incarnations physiques.
Dans
la nature, A, C, G et U / T sont des molécules, stockées sous forme de chaînes
dans l’ADN (ou l’ARN).
Dans
les ordinateurs, on regroupe 8 bits dans un octet, et l’octet est l’unité
typique que l’on utilise pour traiter des données.
La
nature de son côté regroupe 3 nucléotides en un codon, et c’est ce codon qui
est l’unité typique de traitement. Il contient 6 bits d’information (2 bits par
caractère d’ADN, donc 3 caractères = 6 bits. C’est à dire 2⁶ = 64 valeurs différentes possibles pour
un codon).
Très
numérique tout ça ! En cas de doute, allez voir le document de l’OMS avec le
code pour vérifier par vous-même.
Pour
aller plus loin et mieux comprendre le reste de cet article, lisez
celui-ci. Ou si vous préférez les vidéos, en voilà 2 heures pour vous.
Alors, qu’est-ce qu’il fait ce code ?
L’idée
d’un vaccin est d’apprendre à notre système immunitaire comment combattre un
agent pathogène, sans tomber réellement malade.
Historiquement,
on faisait cela en injectant un virus affaibli ou neutralisé (atténué), plus un
«adjuvant» pour réveiller notre système immunitaire. C’était une technique
résolument analogique, qui nécessitait des milliards d’œufs (ou d’insectes).
Cela demandait également beaucoup de chance et beaucoup de temps. Parfois, on
se servait d’un autre virus sans rapport.
Un
vaccin à ARNm atteint le même but («éduquer notre système immunitaire») mais
avec une précision au laser, dans les deux sens du terme : très fin mais aussi
très puissant.
Voici
comment ça fonctionne : l’injection contient du matériel génétique volatil qui
décrit la fameuse protéine «Spike» du SARS-CoV-2 (spicule ou péplomère en
français). Grâce à des astuces chimiques, le vaccin parvient à introduire ce
matériel génétique dans certaines de nos cellules.
Celles-ci
se mettent alors à produire consciencieusement des protéines Spike du
SARS-CoV-2, en quantité suffisante pour que notre système immunitaire entre en
action. Confronté aux protéines Spike et aux signes révélateurs d’une invasion,
il développe alors une réponse puissante contre plusieurs aspects de la
protéine Spike et du processus de production.
Et
c’est comme cela qu’on se retrouve avec un vaccin efficace à 95%.
Le code source !
Commençons
par le commencement. Un schéma dans le document de l’OMS nous aide à comprendre
:

C’est
une sorte de table des matières. Nous allons commencer par la coiffe (“cap”),
représentée par un petit chapeau.
De
la même manière qu’en informatique vous ne pouvez pas juste mettre des opcodes (instructions en langage machine)
nus dans un fichier pour les exécuter, le système d’exploitation biologique
nécessite lui aussi des en-têtes, de l'édition de liens et des choses comme les
conventions d’appel.
Le
code du vaccin commence donc par les deux nucléotides suivants:
GA
On
pourrait comparer cela aux caractères MZ
qui sont au début de tous les exécutables DOS et Windows, ou au #! des
scripts UNIX. Dans les systèmes d’exploitation informatiques comme dans celui
de la vie, ces deux caractères ne sont jamais exécutés mais ils sont
obligatoires, sinon rien ne se passe.
La
coiffe de l’ARNm a un certain nombre de fonctions. D’une part,
elle marque le code comme provenant du noyau de la cellule. Dans notre cas bien
sûr, c’est faux : notre code provient d’une vaccination. Mais ça, la cellule
n’a pas besoin de le savoir. La coiffe donne à notre code une apparence
légitime, qui le préserve de la destruction.
Les
deux nucléotides GA initiaux
sont également légèrement différents chimiquement du reste de l’ARN : c’est une
forme de signalisation hors bande dont ils sont
dotés.
La « région 5' non traduite » (5'-UTR)
Un
peu de jargon maintenant. Les molécules d’ARN ne peuvent être lues que dans une
seule direction. Pour ne pas faire simple, on a nommé l’endroit où la lecture
commence le 5' ou “cinq prime” (nom complet : 5'-UTR, pour Untranslated
Transcribed Region). Et la lecture s’arrête à l’autre extrémité qu’on
appelle 3' ou trois prime (3'-UTR).
La
vie est faite de protéines (ou de choses fabriquées à partir de protéines), et
ces protéines sont décrites par l’ARN. La conversion des instructions de l’ARN
en protéines s’appelle la “traduction”.
Voici
la région “non traduite” 5', qui ne finit donc pas dans la protéine :
GAAΨAAACΨAGΨAΨΨCΨΨCΨGGΨCCCCACAGACΨCAGAGAGAACCCGCCACC
Et
on a ici notre première surprise : les caractères ARN normaux sont A, C, G et U
(Adénine, Cytosine, Guanine et Uracile). U s’appelle aussi «T» (Thymine) dans
l’ADN.
Mais
ici, on trouve à la place “Ψ” : pourquoi ?
C’est
l’une des astuces incroyables de ce vaccin. Notre corps dispose d’un puissant
système antivirus (le premier d’entre tous). Grâce à (ou à cause de) lui, les
cellules sont très méfiantes vis à vis de l’ARN étranger et font tout leur
possible pour le détruire avant qu’il ne puisse faire quoi que ce soit.
Ca
pose évidemment un problème pour notre vaccin : il faut qu’il puisse se frayer
un chemin à travers nos défenses immunitaires. Or, au fil d’années
d’expérimentations, on a découvert que si le U dans l’ARN est remplacé par une
molécule légèrement modifiée, notre système immunitaire le laisse complètement
tranquille. Oui, sérieusement !
Donc
dans le vaccin BioNTech / Pfizer, chaque U a été remplacé par du
1-méthyl-3'-pseudouridylyle, noté Ψ. Ce qui est vraiment fort c’est que,
même si cette substitution permet de ne pas alerter notre système immunitaire,
le Ψ est accepté comme un U normal par les mécanismes cellulaires.
En
sécurité informatique, on connaît bien cette astuce : il est parfois possible
de faire passer une version légèrement altérée d’un message qui va traverser
les pare-feu et systèmes de sécurité, mais qui est quand même acceptée par les serveurs
backend - qui peuvent alors être piratés.
Nous
récoltons aujourd’hui les fruits d’années de recherche fondamentale ayant
permis des découvertes comme celle-ci. Lesinventeurs de cette technique Ψ ont
dû se battre pour que leur travailsoit financé, puis accepté. Nous
devrions tous leur en être très reconnaissants, et je suis sûr que les prix Nobel ne tarderont pas.
Beaucoup
de gens ont demandé : est-ce que les virus pourraient eux aussi utiliser la
technique Ψ pour passer outre nos défenses immunitaires ? Pour faire
court, c’est extrêmement improbable. La vie n’a tout simplement pas de machine
pour fabriquer des nucléotides 1-méthyl-3'-pseudouridylyle. Les virus reposent
entièrement sur les mécanismes de la vie pour se reproduire, et un tel
dispositif n’existe pas. Les vaccins ARNm se dégradent rapidement dans le corps
humain, et il n’est pas possible que l’ARN Ψ-modifié se réplique avec le
Ψ toujours dedans. Une bonne lecture pour aller plus loin : “No, Really, mRNA
Vaccines Are Not Going To Affect Your DNA“
Ok,
revenons au 5'-UTR. A quoi servent ces 51 caractères ? Comme toujours dans la
nature, presque rien n’a une seule fonction bien précise.
Lorsque
nos cellules doivent traduire l’ARN en protéines, elles
utilisent une machine appelée ribosome. Le ribosome est comme une imprimante 3D
à protéines : il ingère un brin d’ARN, et à partir de là émet une chaîne
d’acides aminés, qui se replient ensuite en une protéine.
Source: Wikipedia
C’est
ce que nous voyons ci-dessus. Le ruban noir en bas c’est l’ARN. Le ruban
apparaissant dans le morceau vert est la protéine en cours de formation. Les
choses qui entrent et sortent sont des acides aminés, et des adaptateurs pour
les faire tenir sur l’ARN.
Ce
ribosome doit être physiquement “assis” sur le brin d’ARN pour commencer le
travail. Une fois installé, il peut commencer à former des protéines en
fonction de l’ARN qu’il ingère. Il est donc facile de comprendre qu’il ne peut
pas lire les parties sur lesquelles il atterrit en premier. C’est l’une des
raisons d’être de la séquence UTR: une zone d’atterrissage des ribosomes, qui
fournit une «introduction».
En
plus de cela, la séquence UTR contient des métadonnées: quand la traduction
doit-elle avoir lieu ? En quelle quantité ? Pour le vaccin, ils ont déniché un
UTR qui disait le plus clairement possible «maintenant!», tiré du gène de l'alpha globine. Ce gène est connu pour produire
beaucoup de protéines de manière fiable. Mais ces dernières années, les
scientifiques ont trouvé des moyens d’optimiser encore plus cet UTR (d’après le
document de l’OMS). Il ne s’agit donc pas tout à fait de la séquence UTR de
l’alpha globine : c’est une version améliorée.
Le peptide signal de la glycoprotéine S
Comme
nous l’avons vu, le but du vaccin est d’amener la cellule à produire la
protéine Spike du SARS-CoV-2 en grande quantité. Jusqu’ici, nous avons surtout
vu dans le code source du vaccin des métadonnées et autres éléments accessoires
de «convention d’appel». Maintenant, entrons dans le vif du sujet avec la
partie “protéines virales” proprement dite.
Il
nous reste juste une dernière couche de métadonnées. Une fois que le ribosome
(dans la splendide animation ci-dessus) a fabriqué une protéine, cette protéine
doit aller quelque part. Cette information est encodée dans le «peptide signal
de la glycoprotéine S (séquence de tête étendue)».
Une
manière de voir cela est qu’au début de la protéine il y a une sorte
d’étiquette d’adresse, encodée dans la protéine elle-même. Dans ce cas précis,
le peptide signal dit que cette protéine doit sortir de la cellule via le
«réticulum endoplasmique». Même le jargon de Star Trek n’est pas aussi classe !
Le
«peptide signal» n’est pas très long, mais si on regarde le code, on remarque
des différences entre l’ARN viral et vaccinal (pour faciliter la comparaison,
j’ai remplacé le Ψ modifié par un U normal d’ARN) :
3
3 3 3
3 3 3
3 3 3
3 3 3
3 3 3
Virus:
AUG UUU GUU UUU CUU GUU UUA UUG CCA CUA GUC UCU AGU CAG UGU GUU
Vaccin:
AUG UUC GUG UUC CUG GUG CUG CUG CCU CUG GUG UCC AGC CAG UGU GUU
! !
! ! ! ! ! !
! ! !
! !
Alors
que se passe-t-il ? Je n’ai pas regroupé l’ARN par groupe de 3 lettres au
hasard. Trois caractères ARN, c’est un codon. Et chaque codon code pour un
acide aminé bien précis. Et le peptide signal du vaccin se compose exactement des
mêmes acides aminés que dans le virus lui-même.
Mais
alors, comment se fait-il que l’ARN soit légèrement différent ?
Il
y a 4³ = 64 codons différents, car il y a 4 caractères ARN, et il y en a 3 par
codon. Pourtant, il n’existe que 20 acides aminés différents. Cela signifie que
plusieurs codons codent pour le même acide aminé.
En
fait, la vie utilise une table de correspondance presque universelle entre les
codons d’ARN et les acides aminés:
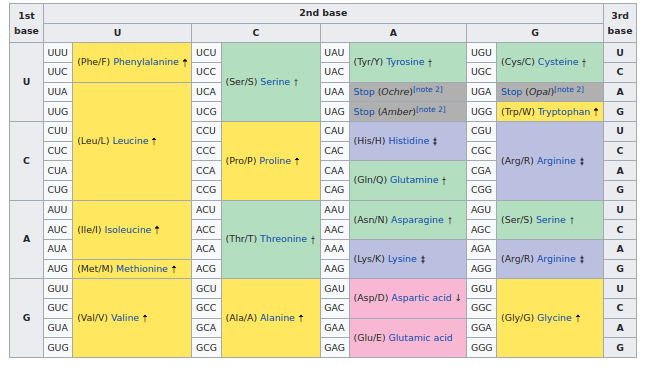
La table des codons ARN (Wikipedia)
Dans
cette table, nous voyons que les modifications du vaccin (UUU -> UUC) sont
toutes synonymes. Le code ARN du vaccin est certes différent, mais
les mêmes acides aminés et la même protéine en sortent.
Si
on regarde de plus près, on voit que la plupart des changements se font à la
troisième position du codon, notée par un «3» ci-dessus. Et si on consulte la
table universelle des codons, on constate que cette troisième position n’a
généralement pas d’impact sur l’acide aminé qui sera produit.
Donc
ces changements sont synonymes, mais alors à quoi servent-ils ? En regardant de
plus près, on remarque que tous sauf un conduisent à plus de C
et de G.
Quel
intérêt ? Comme on l’a vu, notre système immunitaire n’est pas fan d’ARN
«exogène», c’est à dire provenant de l’extérieur de la cellule. C’est pourquoi
on a remplacé le «U» dans l’ARN par un Ψ, pour échapper à la détection.
Mais
il s’avère également que de l’ARN avec une
plus grande quantitéde G et de C est converti plus efficacement en protéines,
C’est
pour cette simple raison que l’ARN du vaccin remplace de nombreux caractères
par des G et des C, partout où c’est possible.
Je
suis assez intrigué par le seul changement qui n’a pas apporté un C ou un G en
plus : la modification CCA -> CCU. Si quelqu’un en connaît la raison,
faites-le moi savoir ! Je sais que certains codons sont plus courants que
d’autres dans le génome humain, mais j’ai aussi lu que ça n’influence pas beaucoup la vitesse
de traduction.
La protéine “Spike” proprement dite
Les
3777 caractères suivants de l’ARN du vaccin ont eux aussi des « codons
optimisés » (avec beaucoup plus de C et de G).
Pour
gagner de la place, je ne vais pas lister tout le code ici, mais nous allons
nous arrêter sur une partie particulièrement intéressante. C’est la portion qui
fait que ça marche, la partie qui nous aidera réellement à revenir à la vie
“normale” :
* *
L
D K V
E A E
V Q I
D R L
I T G
Virus: CUU GAC AAA
GUU GAG GCU GAA GUG CAA AUU GAU AGG UUG AUC ACA GGC
Vaccin: CUG GAC CCU CCU GAG GCC GAG GUG CAG AUC GAC AGA CUG AUC ACA GGC
L
D P P E
A E V
Q I D
R L I
T G
! !!! !! !
! ! !
! ! !
On
retrouve d’abord ici les substitutions habituelles entre synonymes ARN. Par
exemple, dans le premier codon on voit que CUU est remplacé par CUG. Cela
rajoute un «G» au vaccin, ce qui on le sait contribue à améliorer la production
de protéines. CUU et CUG codent tous les deux pour l’acide aminé «L» ou
Leucine, donc rien ne change dans la protéine.
Si
l’on passe en revue la totalité de la protéine Spike dans le vaccin, tous les
remplacements qui ont été faits sont des synonymes comme celui-ci… sauf pour
deux d’entre eux.
Les
troisième et quatrième codons ci-dessus représentent eux de vraies
modifications. Les acides aminés K et V y sont tous les deux remplacés par «P»
ou Proline. Pour ‘K’, il a fallu trois changements ('!!!') et pour «V», il n’en
a fallu que deux ('!!').
Il
se trouve que ces deux remplacements améliorent considérablement l’efficacité
du vaccin.
Par
quel moyen ? Si vous regardez une vraie particule de SARS-CoV-2, vous pouvez
voir la protéine Spike comme un gros tas de spicules (pointes):
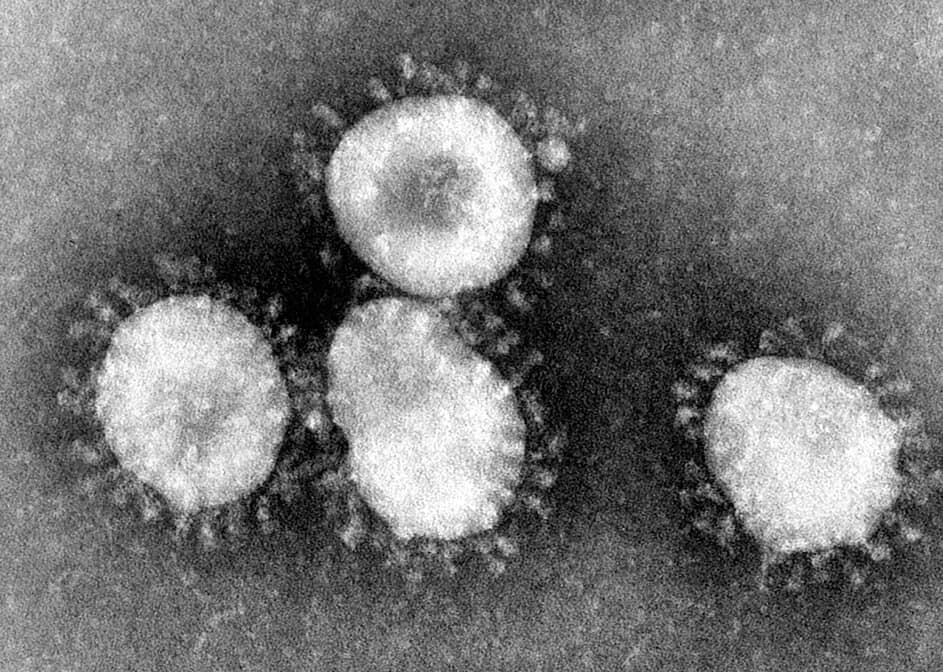
Particules de virus SARS (Wikipedia)
Les
spicules sont montées sur le corps du virus (la «protéine nucléocapside»). Le
problème, c’est que notre vaccin ne génère que les spicules elles-mêmes, sans
les monter sur aucune sorte de corps viral.
Or
il se trouve qu’une protéine Spike non modifiée et non montée sur un virus va
avoir tendance à se recroqueviller, pour former une structure différente. Si
elles étaient injectées en l’état comme vaccin, cela créerait bien une
immunité… mais uniquement contre les protéines Spike «recroquevillées», alors
que le vrai SARS-CoV-2 a lui une protéine Spike bien «pointue» : le vaccin ne
fonctionnerait pas très bien, du coup.
Comment
faire alors ? En 2017, des chercheurs ont découvert que mettre une double
substitution de Proline pile au bon endroit permet aux
protéines SARS-CoV-1 et MERS S de reprendre leur configuration “pré-fusion”,
même sans faire partie d’un virus entier. Cela fonctionne parce que la Proline
est un acide aminé très rigide. Elle agit comme une sorte d’attelle,
stabilisant la protéine dans l’état qu’il faut pour la présenter au système
immunitaire !
Les gens qui
ont fait
cette découverte seraient franchement en droit d’embrasser leur
miroir à chaque fois qu’ils se voient dedans. Ils pourraient transpirer la
suffisance à un degré invraisemblable, et ce serait complètement mérité.
Des
nouvelles ! L’auteur a été contacté par le McLellan lab, l’une des équipes qui a fait la
découverte de la Proline. Ils expliquent qu’au niveau fierté, ils gardent la
tête froide à cause de la pandémie, mais qu’ils sont heureux d’avoir contribué
aux vaccins. Ils insistent aussi sur l’importance de beaucoup d’autres équipes,
travailleurs et volontaires.
La fin de la protéine : étapes suivantes
Si
l’on parcourt le reste du code source, on trouve quelques autres petites
modifications à la fin de la protéine Spike:
V L K
G V K
L H Y
T s
Virus:
GUG CUC AAA GGA GUC AAA UUA CAU UAC ACA UAA
Vaccin:
GUG CUG AAG GGC GUG AAA CUG CAC UAC ACA UGA UGA
V L K
G V K
L H Y
T s s
! !
! ! ! !
! !
À
la fin d’une protéine se trouve un codon «stop», noté ici par un «s» minuscule.
C’est une manière polie de dire que la protéine doit s’arrêter là. Le virus
d’origine utilise le codon stop UAA, et le vaccin utilise lui deux codons stop
UGA, peut-être juste pour faire bonne mesure.
La région 3'-UTR
De
la même manière que le ribosome avait besoin d’une introduction à l’extrémité
5' (où nous avons vu la «région 5' non traduite»), on retrouve à la fin d’une
protéine une construction similaire appelée 3'-UTR.
On
pourrait écrire beaucoup de choses sur la 3'-UTR, mais je cite iciWikipédia: «La région 3'-UTR joue un rôle
crucial dans l’expression des gènes en influençant la localisation, la
stabilité, l’exportation et l’efficacité de la traduction d’un ARNm … malgré
notre compréhension actuelle des 3'-UTR, elles restent encore des mystères
relatifs ».
Ce
que nous savons, c’est que certaines 3'-UTR sont très douées pour promouvoir
l’expression des protéines.
Selon
le document de l’OMS, la 3'-UTR du vaccin BioNTech / Pfizer a été choisie à
partir de «l’amplificateur amino-terminal de l’ARNm scindé (AES) et de l’ARN
ribosomal 12S encodé pour les mitochondries, pour conférer une stabilité à
l’ARN et une expression protéique totale élevée».
Moi
je dis juste : bien joué.
LAAAAAAAAAAAAAAAAAAAAAA fin
La
toute fin de l’ARNm est polyadénylée. C’est un mot savant pour dire qu’elle se
finit par beaucoup de AAAAAAAAAAAAAAAAAAA. Même l’ARNm en a marre de 2020, on
dirait.
L’ARNm
peut être réutilisé plusieurs fois, mais lorsque cela se produit, il perd une
partie de ses A à la fin. Quand il n’y a plus de A, l’ARNm n’est plus
fonctionnel et est éliminé. La queue «poly-A» est donc une protection contre la
dégradation.
Des
études ont été menées pour savoir quel est le nombre optimal de A à la fin pour
les vaccins à ARNm. J’ai lu dans la littérature que ce chiffre est d’environ
120.
Le
vaccin BNT162b2 se termine par:
******
****
UAGCAAAAAA AAAAAAAAAA AAAAAAAAAA
AAAAGCAUAU GACUAAAAAA AAAAAAAAAA
AAAAAAAAAA AAAAAAAAAA AAAAAAAAAA
AAAAAAAAAA AAAAAAAAAA AAAA
Cela
fait 30 A, puis un “linker” (site de liaison) de 10 nucléotides (GCAUAUGACU),
suivi à nouveau de 70 A.
Je
suppose que ce que nous voyons ici est le résultat d’une optimisation
propriétaire plus poussée, pour améliorer encore plus l’expression des
protéines.
En résumé
Nous
connaissons maintenant le contenu exact de l’ARNm du vaccin BNT162b2, et nous
comprenons le rôle de la plupart des composants :
- La coiffe (CAP) pour s’assurer que l’ARN
ressemble à de l’ARNm normal
- Une région non traduite (UTR) 5' dont on sait
qu’elle marche, et qu’on a optimisée.
- Un peptide signal aux codons optimisés, pour envoyer
la protéine Spike au bon endroit (copie conforme à 100% de celui du virus)
- Une version “codons optimisés” de la protéine
Spike d’origine, avec deux substitutions “Proline” pour s’assurer que la
protéine apparaît sous la bonne forme.
- Une région non traduite (UTR) 3' dont on sait
qu’elle marche, elle aussi optimisée.
- Une queue poly-A quelque peu mystérieuse, avec un
“linker” (site de liaison) inexpliqué au milieu.
L’optimisation
des codons ajoute beaucoup de G et C à l’ARNm. Dans le même temps, l’utilisation
du Ψ (1-méthyl-3'-pseudouridylyle) au lieu de U aide à échapper à la
vigilance de notre système immunitaire, afin que l’ARNm reste présent assez
longtemps pour pouvoir réellement remplir sa mission.
Pour aller plus loin
En
2017 l’auteur a fait une présentation de 2 heures sur l’ADN, visible ici.
Comme pour cet article, la cible est un public d’informaticiens.
Il
maintient également une page “l’ADN pour les développeurs” depuis
2001.
Pour
finir, ses
billets de blog contiennent de nombreuses informations sur
l’ADN, SARS-CoV-2 et COVID-19.
Vous
pouvez continuer votre lecture avec la partie 2 qui lance un défi
d’optimisation de codons aux développeurs !
Partie 2 : Explorons le code source du vaccin BioNTech / Pfizer contre le
SARS-CoV-2
January 1,
2021
Toutes
les données sur le vaccin BNT162b2 de cette page proviennent de ce document de l’Organisation mondiale de la Santé.
Cet article est la suite d’une première partie qui explore le
fonctionnement du vaccin BioNTech / Pfizer contre le SARS-CoV-2. Commencez par
la lire si vous ne l’avez pas encore fait !
Ici on va s’intéresser particulièrement à
l’optimisation des codons, et lancer un challenge aux développeurs pour la
création d’un algorithme qui produit un résultat le plus proche possible de
l’optimisation manuelle du vaccin BioNTech.
Pour le tableau des scores du challenge et les mises à
jour, rendez-vous sur l’article original en anglais de Bert Hubert
Traduction par Renaud
Guérin
En
bref: l’ARNm du vaccin a été optimisé par le fabricant grâce à des
substitutions dans l’ARN (par exemple, remplacer UUU par UUC)
et les gens aimeraient comprendre la logique derrière ces modifications.
Il
s’agit d’un problème assez proche de ceux que les cryptologues et les hackers
qui décompilent du code rencontrent au quotidien. Sur cette page, vous
trouverez tous les détails dont vous avez besoin pour faire l’ingénierie
inverse du code du vaccin et comprendre comment il a été optimisé.
Au
départ je pensais que ce serait juste un casse-tête amusant, mais je viens
d’apprendre que trouver un bon algorithme d’optimisation et le documenter est
en fait extrêmement important pour les chercheurs, et pourrait les aider à
concevoir du code pour les protéines et les vaccins.
Donc,
si vous voulez aider à la recherche sur les vaccins, poursuivez votre lecture !
Le tableau des scores
Chacun est invité à envoyer son algorithme à l’auteur
surbert@hubertnet.nl ou @PowerDNS_Bert.
Le tableau des meilleurs scores n’est pas dans cette
traduction, retrouvez-le dans l’article original
BioNTech
C’est
une chance que BioNTech ait partagé ces données publiquement. Et bien sûr, nous
devons beaucoup aux nombreux chercheurs qui ont travaillé pendant des décennies
pour faire avancer la science au point de rendre possible un tel vaccin ! C’est
véritablement une merveille.
Et
c’est parce que c’est une merveille que je veux tout comprendre sur ce vaccin.
La partie 1 décrit en détail ce que contient l’ARNm du vaccin.
Cela vous aidera de la lire avant de continuer, je vous promets que ça en vaut
la peine.
Cela
dit, cette première partie a laissé certaines questions sans réponses, et c’est
là que cela devient passionnant.
L’optimisation des codons
Le
vaccin contient du code ARN qui crée une copie très légèrementmodifiée
de la protéine S du SARS-CoV-2.
Pourtant,
le code ARN du vaccin en lui-même est très différent de
l’original du virus. Le fabricant a fait ces modifications en se basant sur sa
compréhension de la nature.
Et
de ce que nous avons compris, ces modifications rendent le vaccin beaucoup plus
efficace : ce serait donc très intéressant de les regarder dans le détail.
Elles pourraient par exemple expliquer pourquoi le vaccin Moderna a besoin de
100 microgrammes et le vaccin BioNTech seulement 30 microgrammes.
Voici
le début de la protéine S dans le code ARN du virus et dans celui du vaccin
BNT162b2. Les points d’exclamation indiquent les différences.
Virus: AUG UUU GUU UUU CUU GUU UUA
UUG CCA CUA GUC UCU AGU CAG UGU GUU
Vaccin: AUG UUC GUG UUC CUG GUG CUG
CUG CCU CUG GUG UCC AGC CAG UGU GUU
! !
! ! ! ! ! !
! ! !
! !
L’ARN
est littéralement une chaîne de caractères A, C, G et U.
Ces nucléotides ne sont pas physiquement groupés entre eux, mais quand on les
analyse de manière logique, cela a du sens de les regarder comme des groupes de
3.
Chaque
groupe (appelé codon) correspond à un acide aminé (indiqué par une lettre
majuscule). Une chaîne d’acides aminés est une protéine. Voici à quoi ça
ressemble:
Virus: AUG UUU GUU UUU CUU GUU UUA
UUG CCA CUA GUC UCU AGU CAG UGU GUU
M F V
F L V L L
P L V
S S Q
C V
Vaccin: AUG UUC GUG UUC CUG GUG CUG CUG CCU CUG GUG
UCC AGC CAG UGU GUU
!
! ! ! !
! ! ! ! !
! ! !
On
voit bien ici que même si les codons sont différents, leur traduction en acides
aminés ne change pas. Il y a 4 x 4 x 4 (64) codons mais seulement 20 acides
aminés. Cela veut dire que pour chaque codon, vous avez généralement 2 autres
substitutions possibles qui coderont toujours pour le même acide aminé.
Ainsi,
dans le deuxième codon, UUU a été remplacé par UUC.
Cela rajoute un C au vaccin. Le troisième codon est
passé de GUU à GUG,
ce qui rajoute un G.
On
sait qu’une proportion plus élevée de caractères G et C améliore
l’efficacité d’un vaccin à ARNm.
S’il
n’y avait que ça à faire, on pourrait s’arrêter là : «L’algorithme consiste à
remplacer les codons pour avoir un maximum de G et de C».
Mais
c’est là qu’on remarque le 9ème codon, qui lui remplace CCA enCCU.
Au cours des 4000 caractères et quelques du vaccin, cela se produit plusieurs
fois.
Notre défi
Le
but : trouver un algorithme qui transforme le code ARN “sauvage” en celui du
vaccin BNT162b2, parce que tout le monde voudrait comprendre comment
transformer de l’ARN viral en vaccin efficace.
L’algorithme
ne doit pas forcément générer exactement le même code ARN,
mais ce serait super s’il produisait quelque chose de très proche, tout en
restant court.
Pour
vous aider, j’ai fourni les données sous plusieurs formes, comme expliqué sur la
page GitHub.
A
noter que dans ces fichiers, le U mentionné
ci-dessus apparaît comme un T. U et T sont
les versions ARN et ADN de la même information.
L’endroit
le plus simple pour démarrer est le fichier ‘side-by-side.csv’. Il contient la version
originale et modifiée de chaque codon, côte à côte:
abspos,codonOrig,codonVaccine
0,ATG,ATG
3,TTT,TTC
6,GTT,GTG
...
3813,TAC,TAC
3816,ACA,ACA
3819,TAA,TGA
Il
y a aussi une table d’équivalence qui montre quels codons peuvent être
interchangés sans affecter les acides aminés produits. Vous la trouverez dans codon-table-grouped.csv. Et une visualisation ici.
Un exemple d’algorithme
Dans
le dépôt GitHub, vous trouverez 3rd-gc.gp.
Il
implémente une stratégie simple qui fonctionne comme ceci:
- Si un codon de virus se terminait déjà par G ou
C, on le copie dans l’ARNm du vaccin.
- Sinon, on remplace le dernier nucléotide du codon
par un G, on regarde si l’acide aminé correspond toujours : si oui, on le
copie dans l’ARNm du vaccin.
- On essaie la même chose avec un C.
- A défaut, on copie tel quel.
// cas par défaut, on ne fait rien
our = vir
// on ne fait rien si le codon se termine déjà par G ou C
if(vir[2] == 'G' || vir[2]
=='C') {
fmt.Printf("Le codon se termine déjà par G ou C,
on ne fait rien.")
} else {
prop = vir[:2]+"G"
fmt.Printf("Tentative de
substitution d'un G, nouveau candidat '%s'. ", prop)
if(c2s[vir] == c2s[prop]) {
fmt.Printf("L'acide
aminé est toujours le même, c'est fait!")
our = prop
} else {
fmt.Printf("Oups,
l'acide aminé a changé. On essaie avec C, nouveau candidat '%s'. ", prop)
prop =
vir[:2]+"C"
if(c2s[vir] ==
c2s[prop]) {
fmt.Printf("L'acide
aminé est toujours le même, c'est fait!")
our=prop
}
}
}
Avec
cet algorithme, on arrive à une correspondance plutôt médiocre de 53,1% avec le
vaccin BioNTech, mais c’est un début !
Lorsque
vous concevrez votre algorithme, assurez-vous de ne vous baser que sur l’ARN du
virus. Ne trichez pas en allant voir l’ARN BioNTech !
Si
vous avez atteint un score supérieur à 53,1%, envoyez un lien vers votre code à bert@hubertnet.nl (ou @PowerDNS_Bert)
et il sera ajouté au tableau des scores !
Astuces pour faciliter la
tâche
Comme
toujours en matière de rétro-ingénierie ou de cryptanalyse, cela aide de
comprendre ce que l’on a sous les yeux.
Proportion de GC
Nous
savons que l’un des objectifs de «l’optimisation des codons» est d’obtenir plus
de C et de G dans la version
vaccin de l’ARN.
Mais
il y a aussi une limite à cela. Dans l’ADN (qui intervient également dans la fabrication
du vaccin) G et C se
lient fortement, au point que si vous mettez trop de ces «nucléotides», il ne
se réplique plus efficacement.
Du
coup, certaines des modifications sont en fait là pour réduire le
pourcentage de GC d’un segment d’ADN s’il commençait à devenir trop élevé.
L’auteur a tweeté à ce sujet.
L’optimisation des codons
Certains
codons sont rares dans l’ADN humain ou dans certaines cellules. Il se peut que
certains codons soient remplacés par d’autres simplement parce qu’ils sont plus
couramment utilisés par certaines cellules. Un autre tweet à ce sujet .
Pliage de l’ARN
Jusqu’ici
nous ne nous sommes intéressés qu’aux codons.
Mais
l’ARN lui-même ne connaît pas le concept de codons : il n’y a pas de marqueur
physique pour indiquer où un codon commence et se termine. Cela dit, le premier
codon d’une protéine est toujours ATG (ou AUG en version ARN).
L’ARN
se recroqueville dans une certaine forme. Il se peut que cette forme aide à
échapper au système immunitaire, ou à améliorer la traduction en acides aminés.
Cela ne dépend que de la séquence de nucléotides ARN, et non de codons
spécifiques.
Vous
pouvez envoyer des séquences ARN à ce serveur de l’Institut de chimie théorique de
l’Université de Vienne et il pliera l’ARN pour vous. C’est un
serveur très sophistiqué qui effectue des calculs méticuleux.
Cette page Wikipédia décrit comment cela
fonctionne.
Il
se peut que certaines optimisations améliorent le pliage.
On
me dit également que cet article de Moderna (un autre fabricant de vaccin à
ARNm) peut être utile: la structure
de l’ARNm régule l’expression des protéines par des changements dans la
demi-vie fonctionnelle.